m = 84, n = 12600, n_c = 799
Scree Plots
| Y | Y_c |
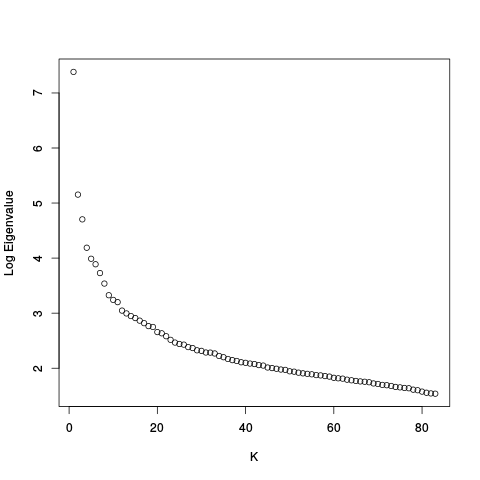 |
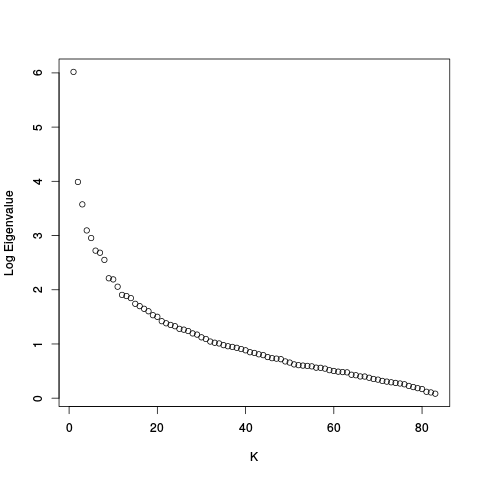 |
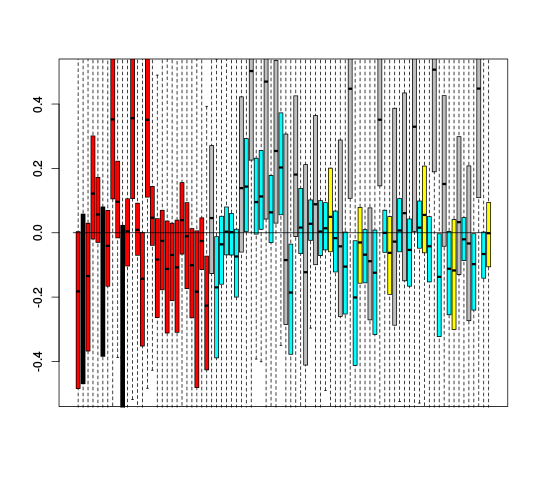
RLE Plots
| Y | Y_c |
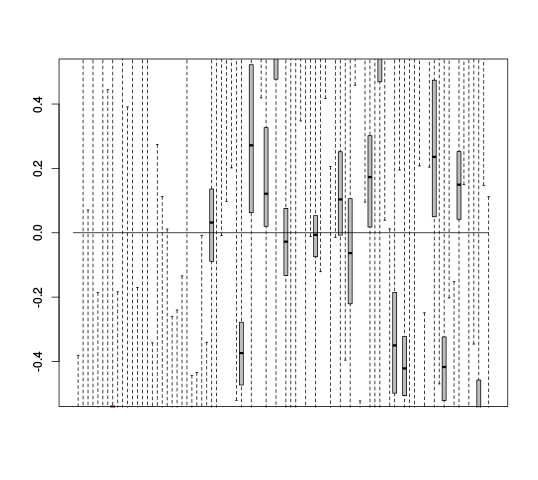 |
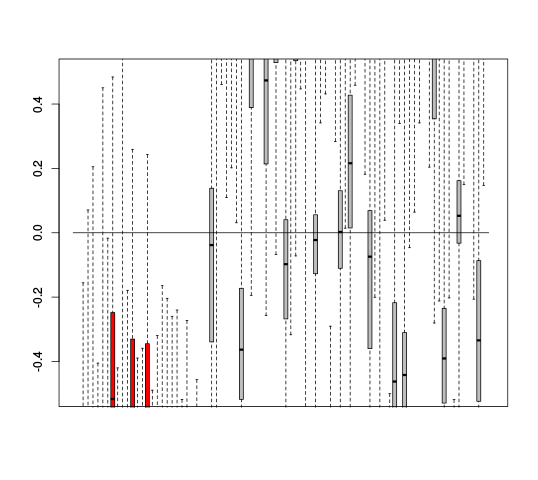 |
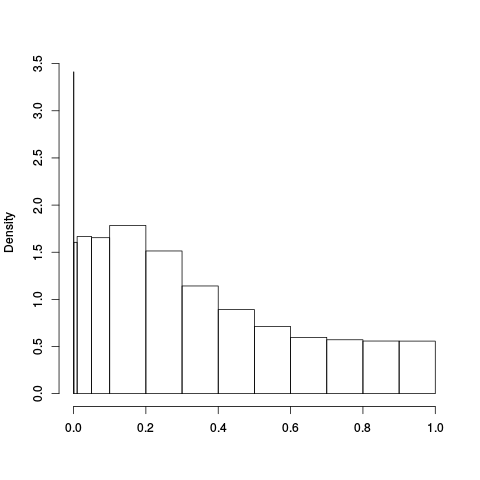
Cancor Plots
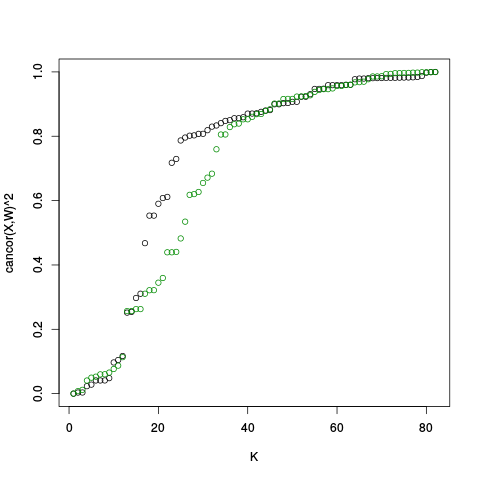 |
PC Plots
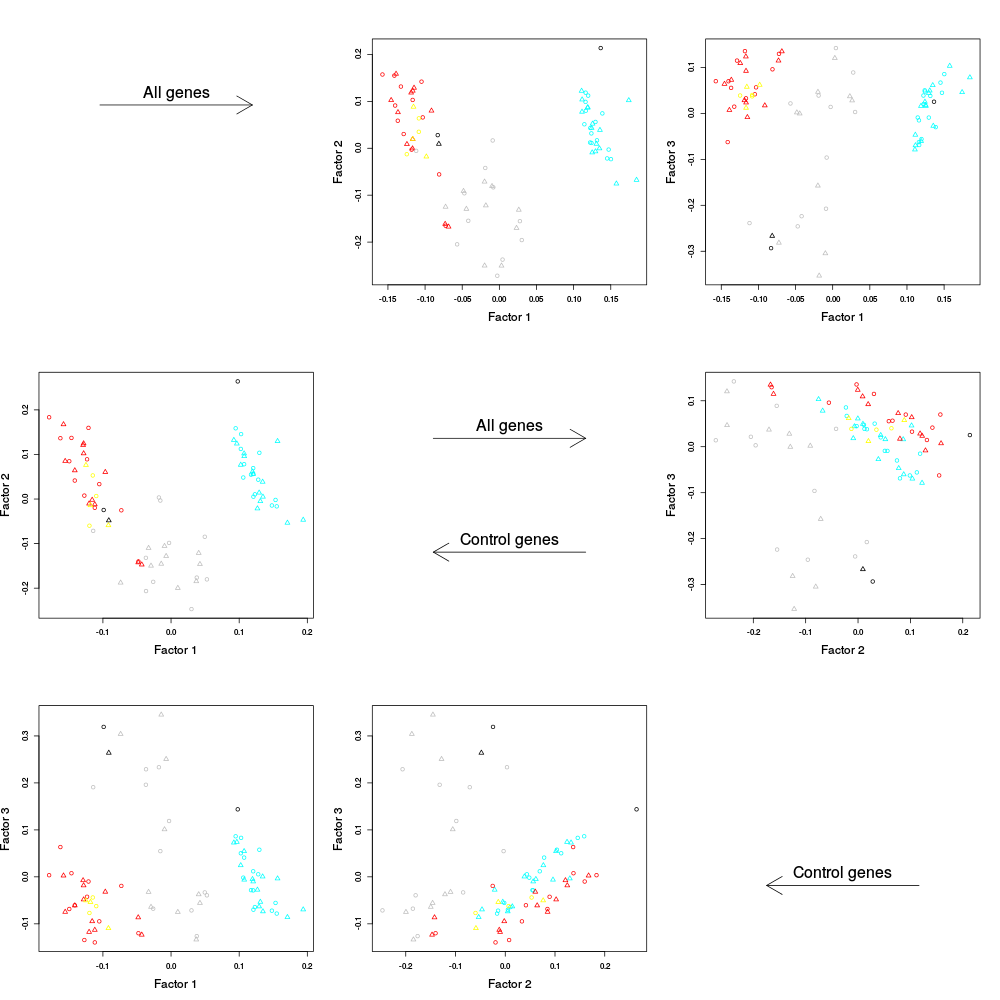 |
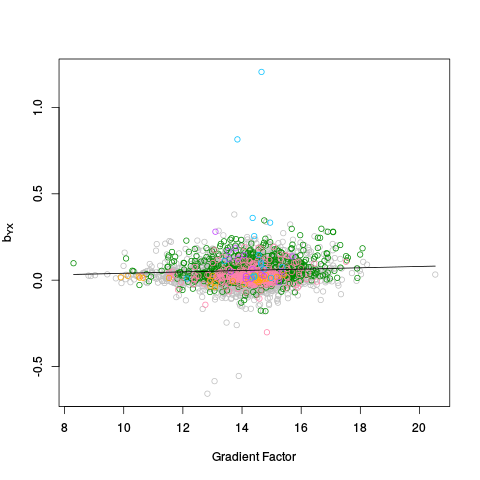
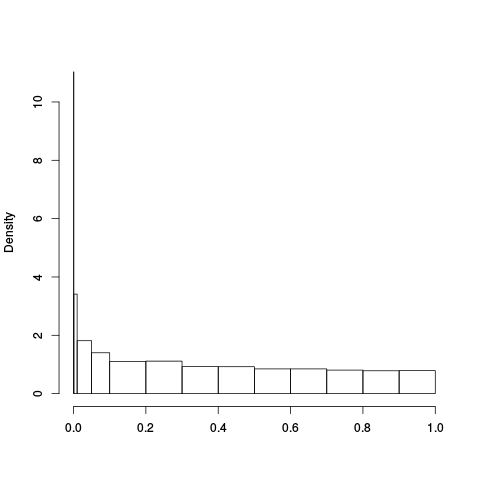
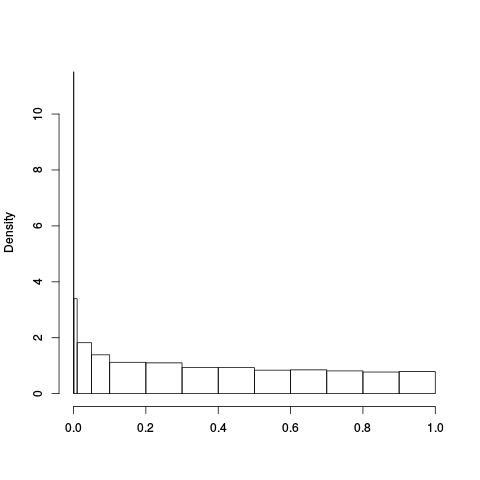
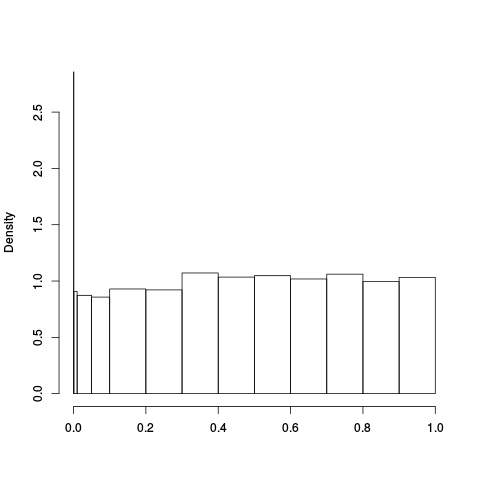
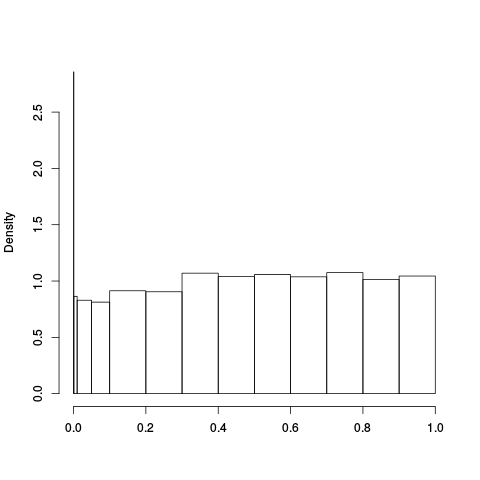
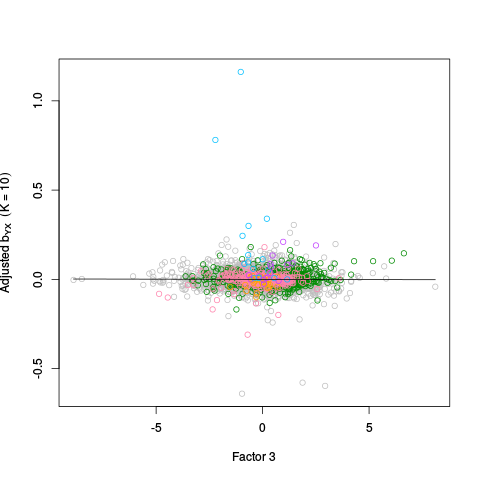
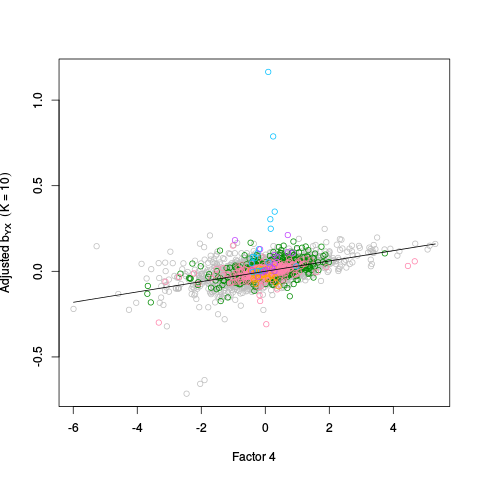
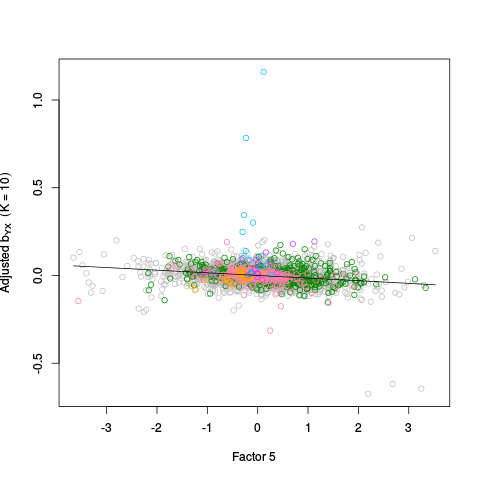
Alpha Plots
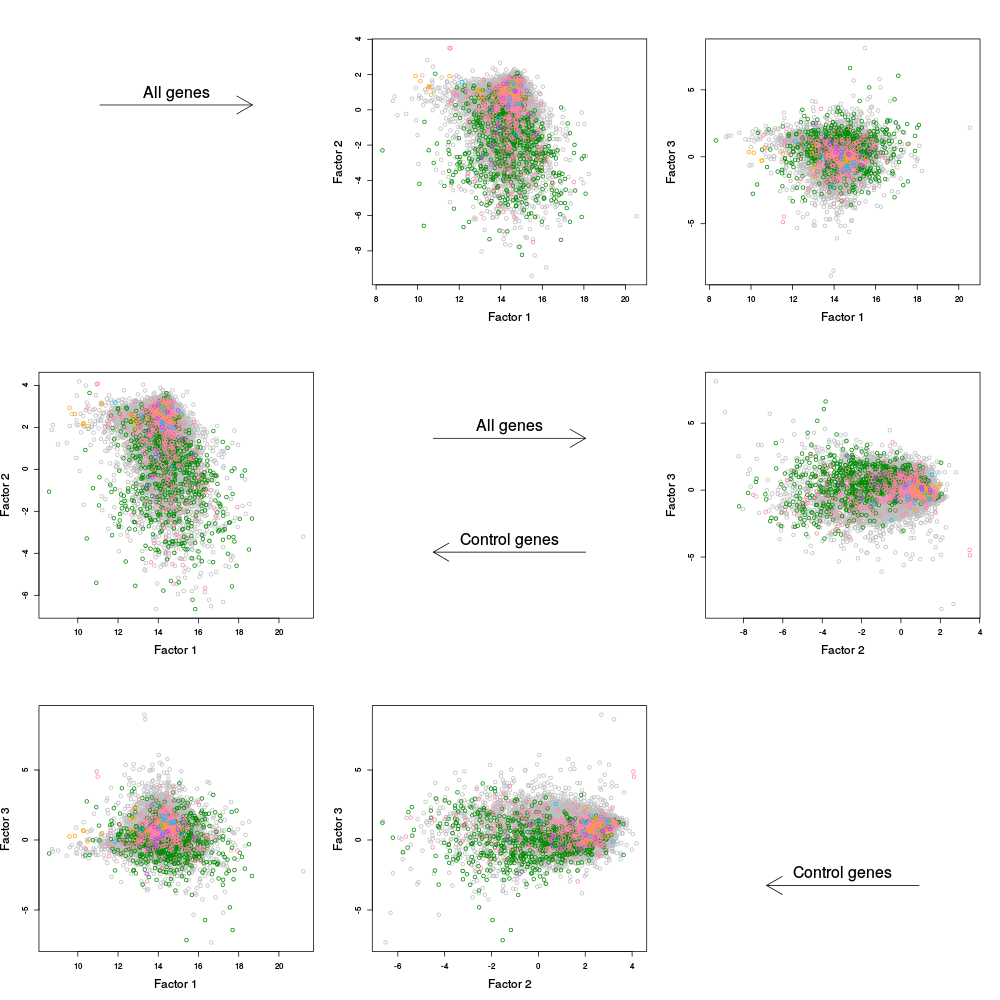 |
| Y | Y_c |
 |
 |
| Y | Y_c |
 |
 |
 |
 |
 |
| standard | ebayes | rsvar | rsvar ebayes | evar |
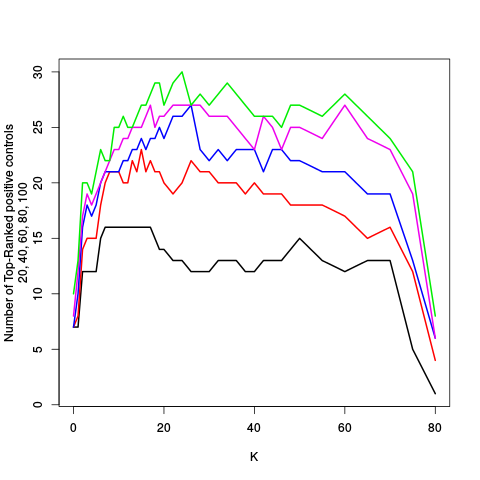 |
 |
 |
 |
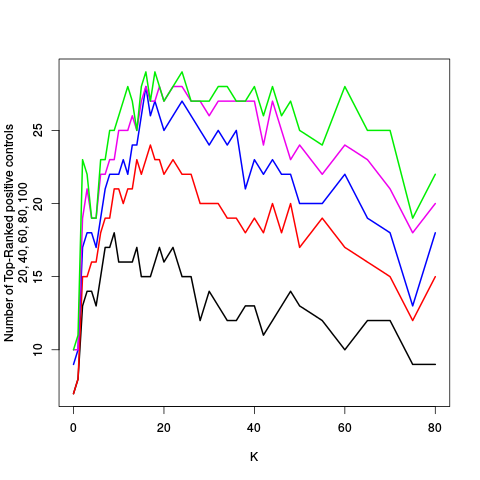 |
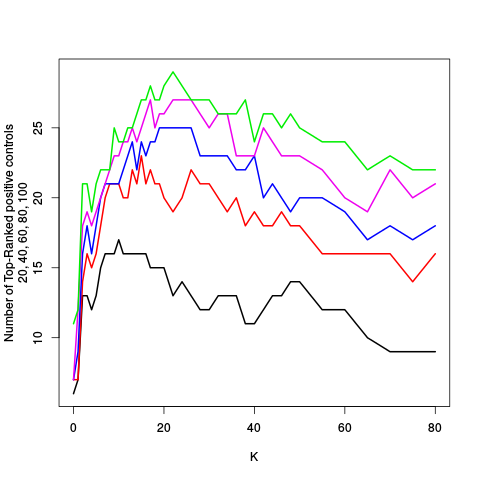
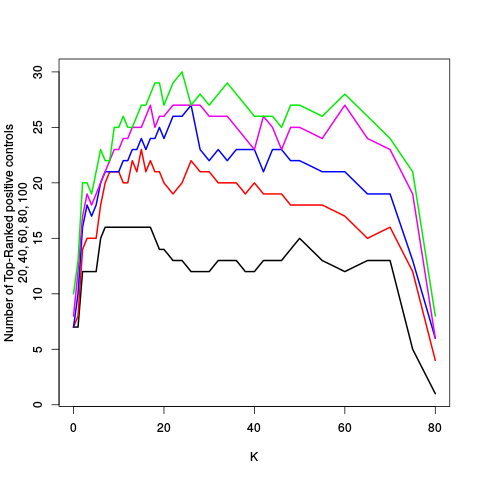
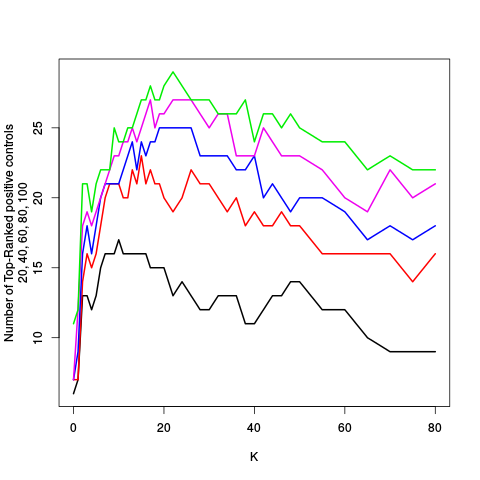
| K | Y | Y_c | projection | standard | ebayes | rsvar | rsvar ebayes | evar | standard | ebayes | rsvar | rsvar ebayes | evar | standard | ebayes | rsvar | rsvar ebayes | evar | standard (controls) | ebayes (controls) | rsvar (controls) | rsvar ebayes (controls) | evar (controls) | standard (controls) | ebayes (controls) | rsvar (controls) | rsvar ebayes (controls) | evar (controls) | standard (controls) | ebayes (controls) | rsvar (controls) | rsvar ebayes (controls) | evar (controls) |
| 1 | 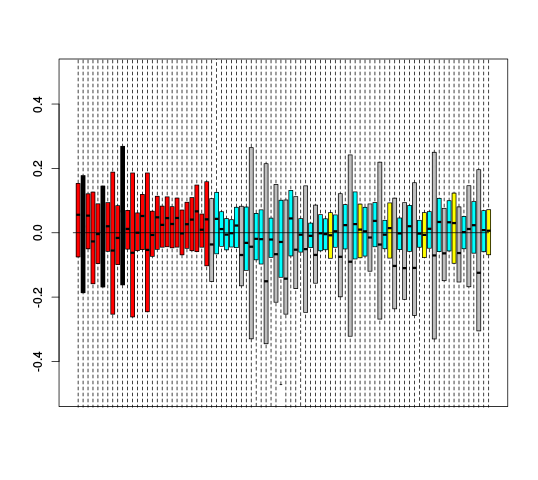 |
 |
 |
 |
 |
 |
 |
 |
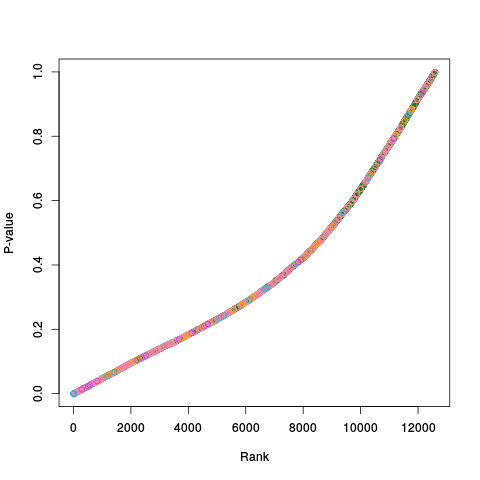 |
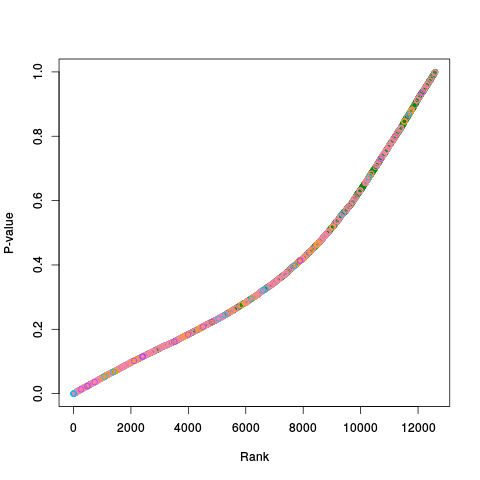 |
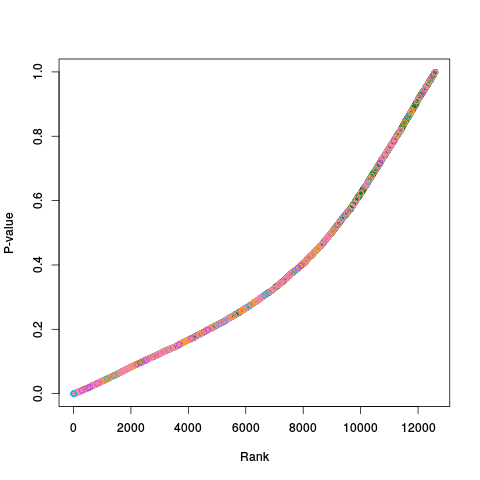 |
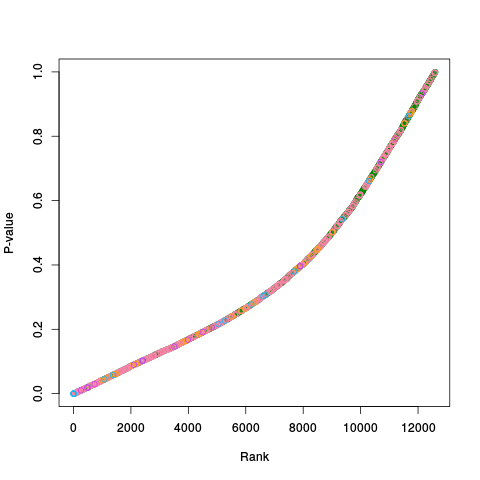 |
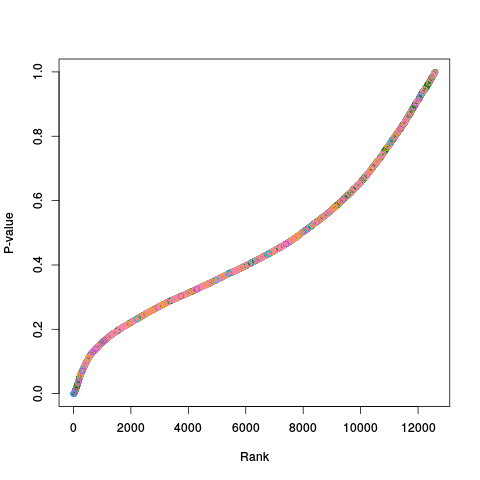 |
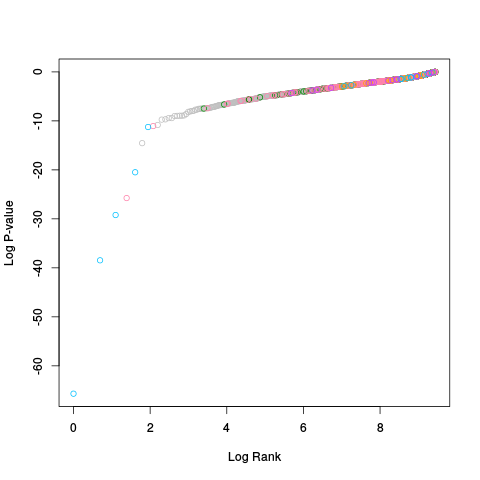 |
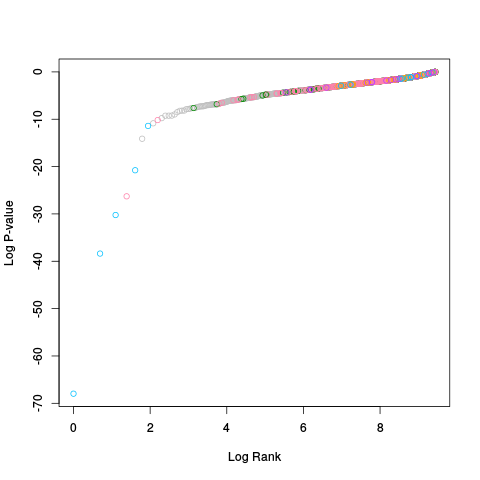 |
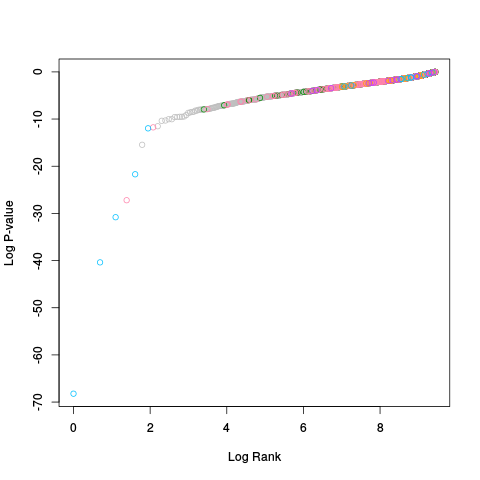 |
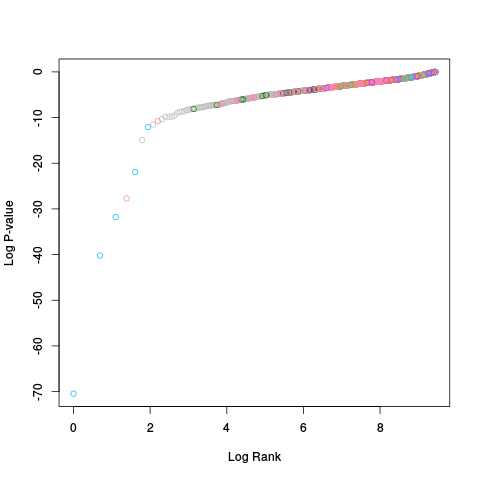 |
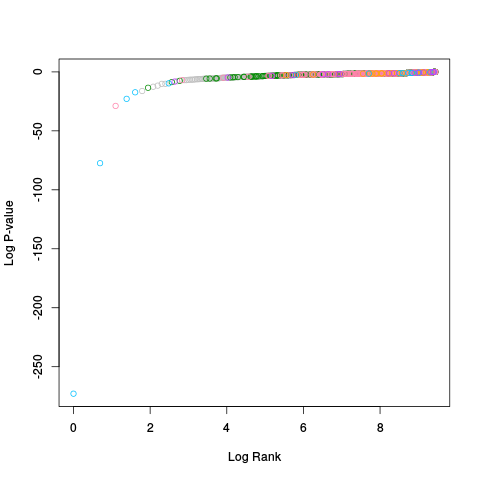 |
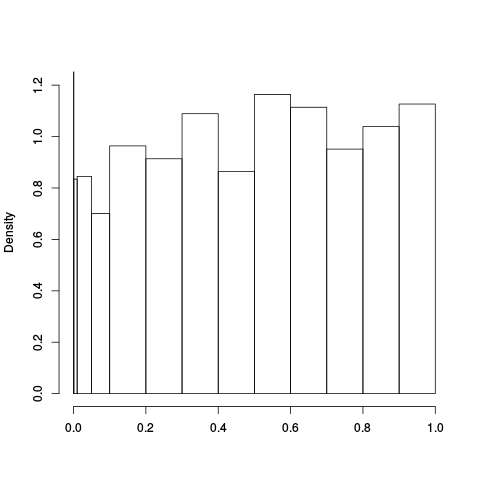 |
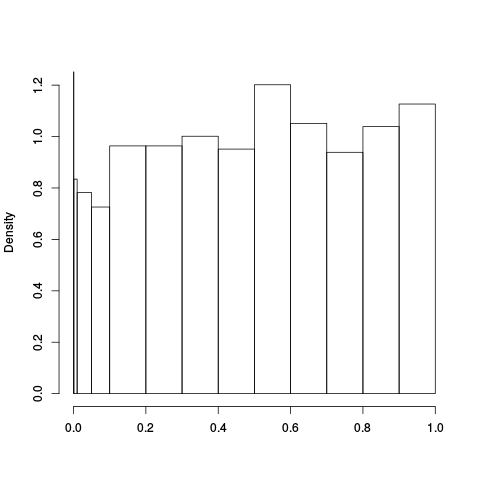 |
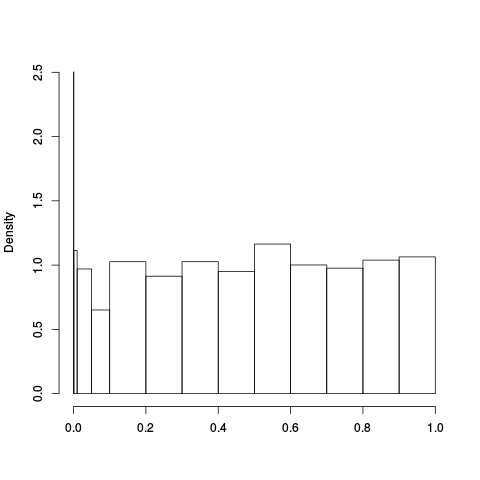 |
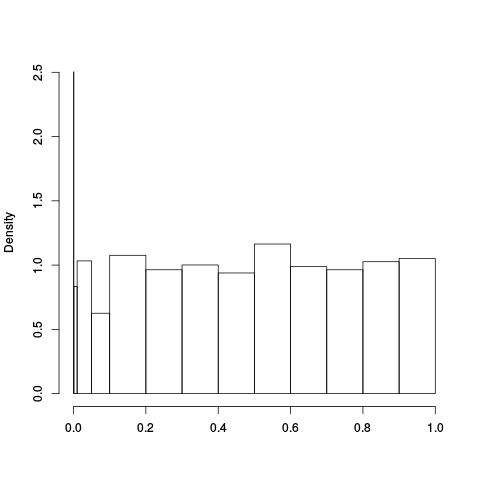 |
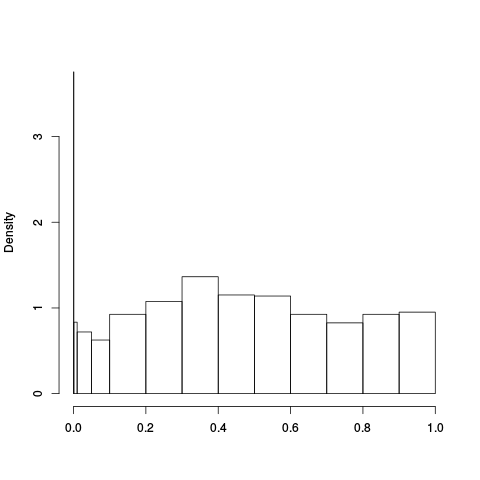 |
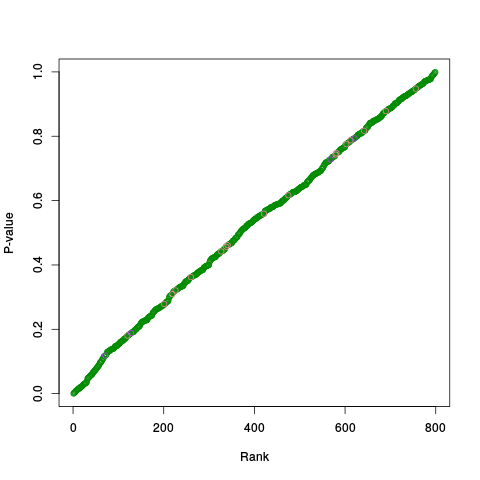 |
 |
 |
 |
 |
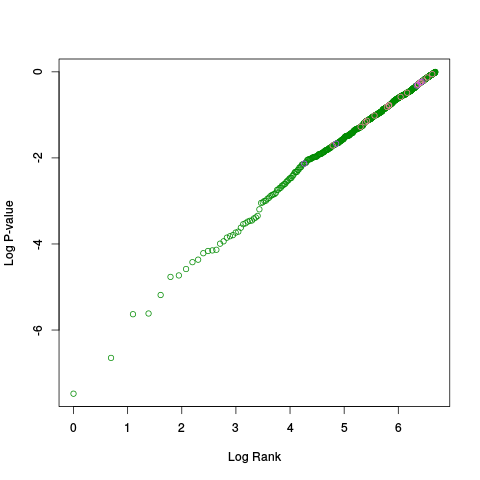 |
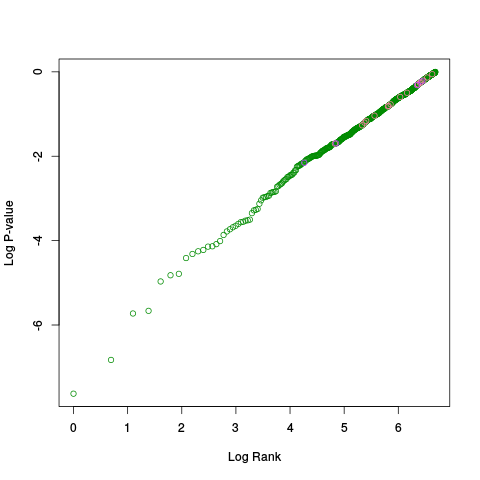 |
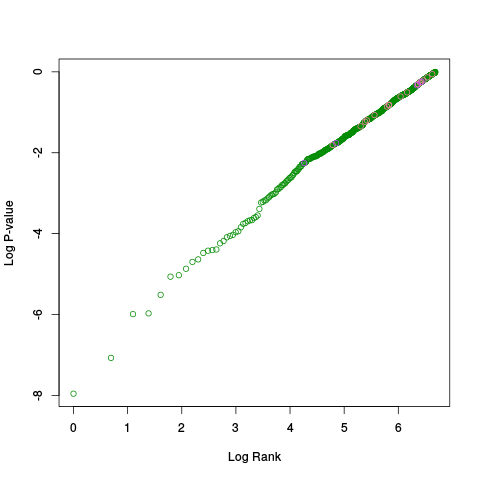 |
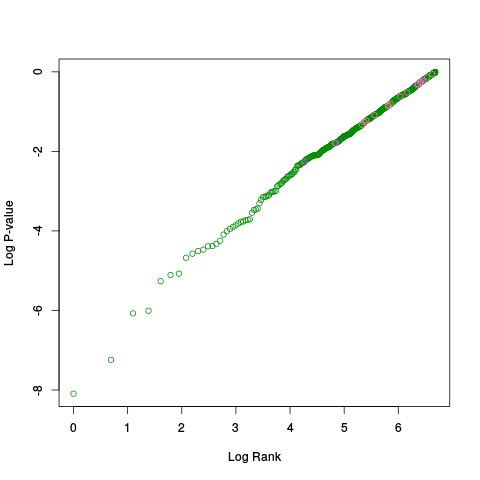 |
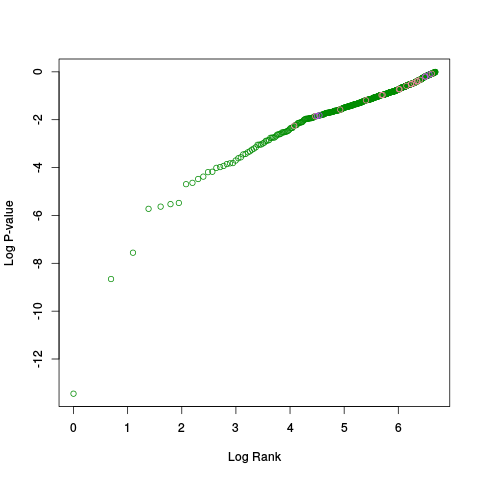 |
| 10 | 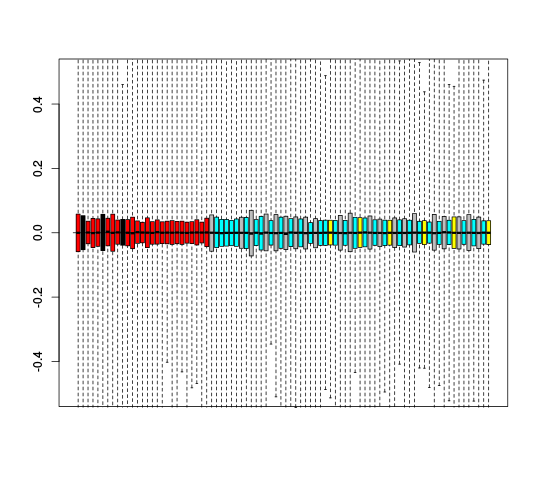 |
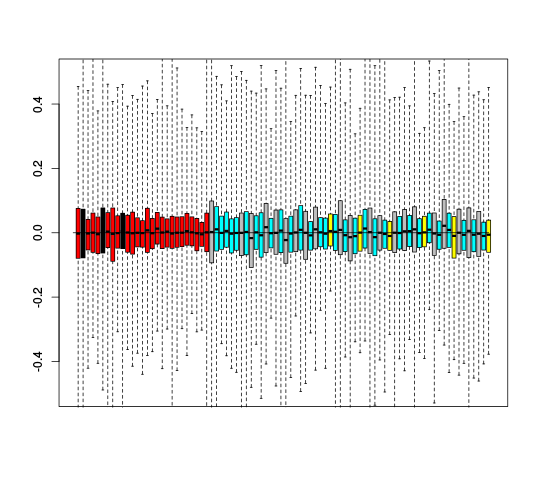 |
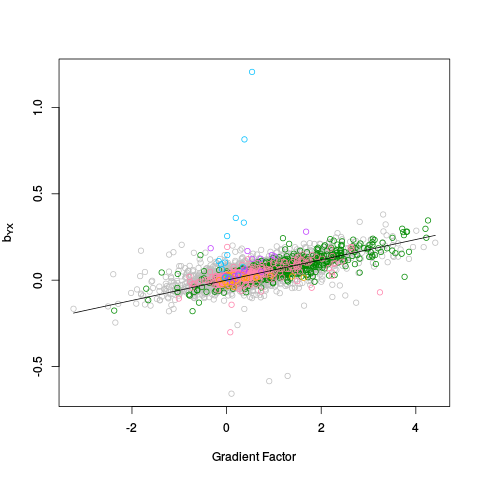 |
 |
 |
 |
 |
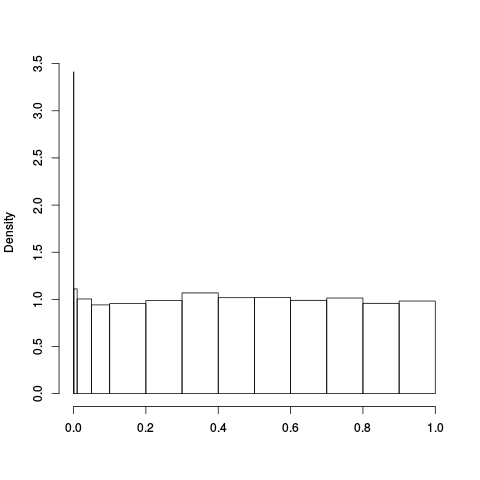 |
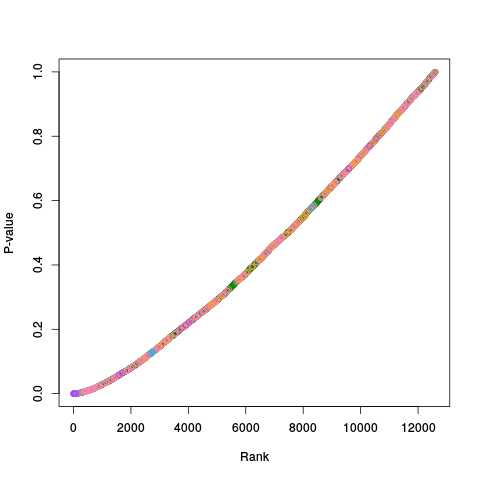 |
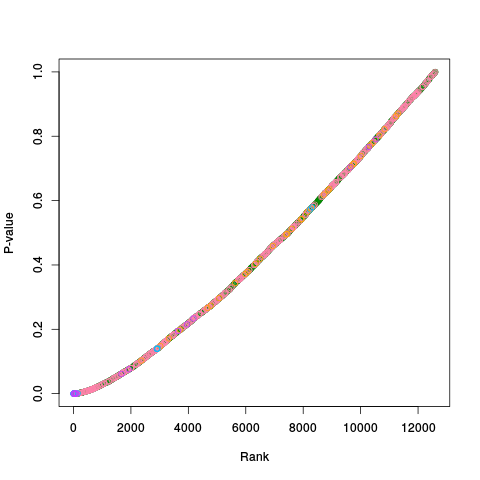 |
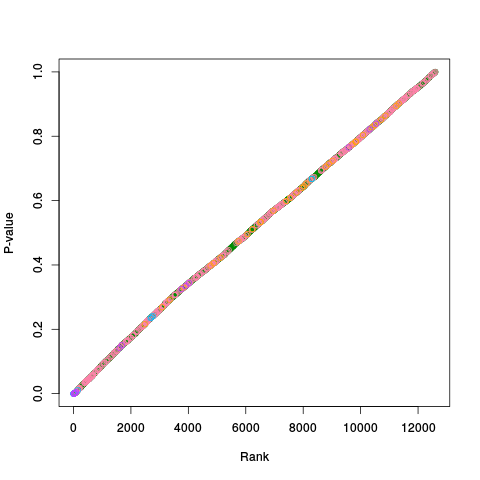 |
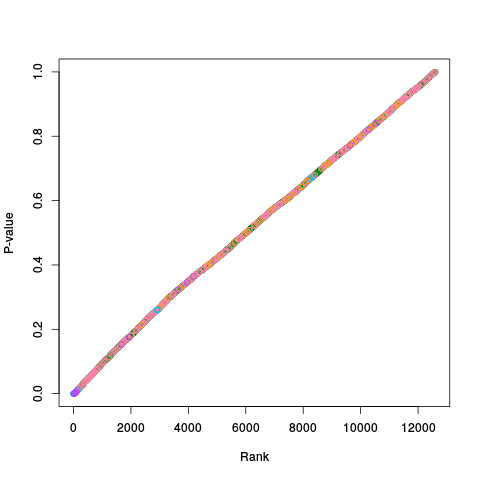 |
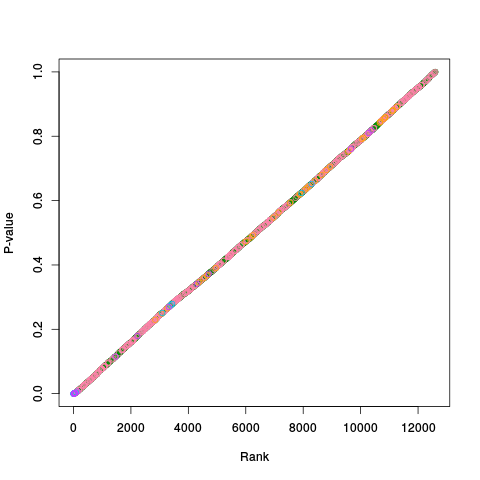 |
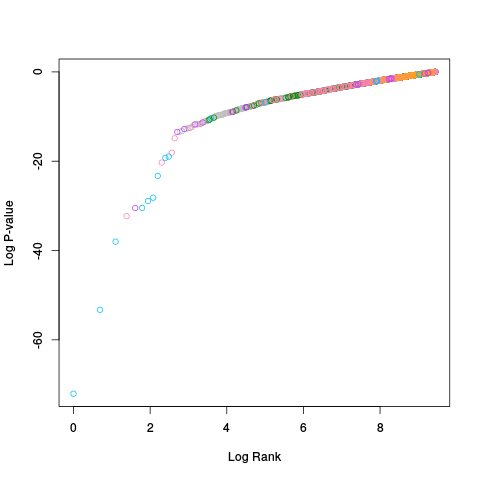 |
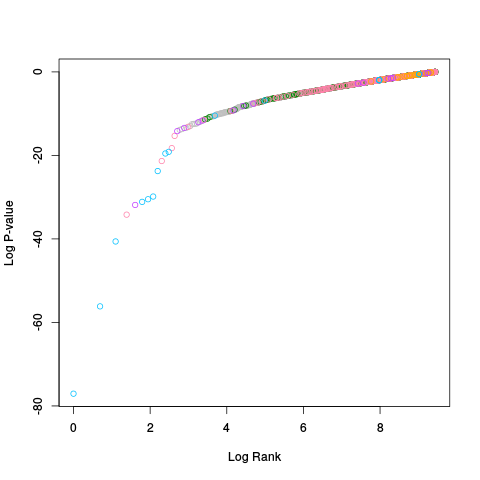 |
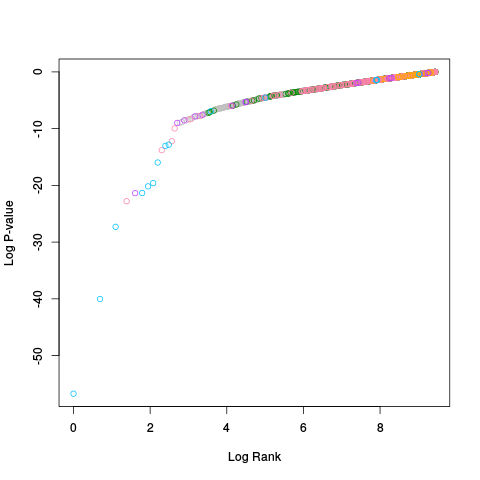 |
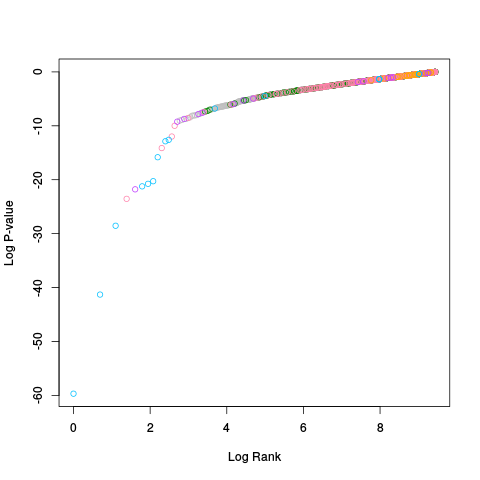 |
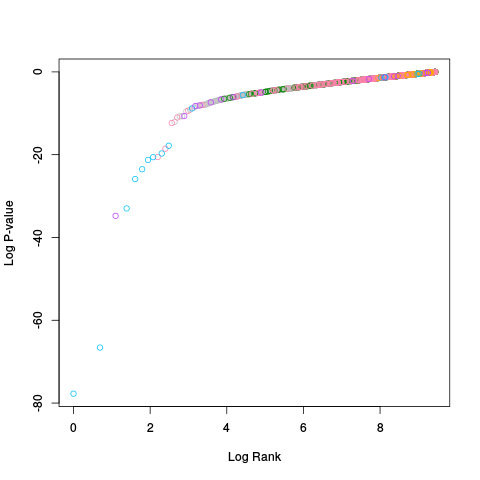 |
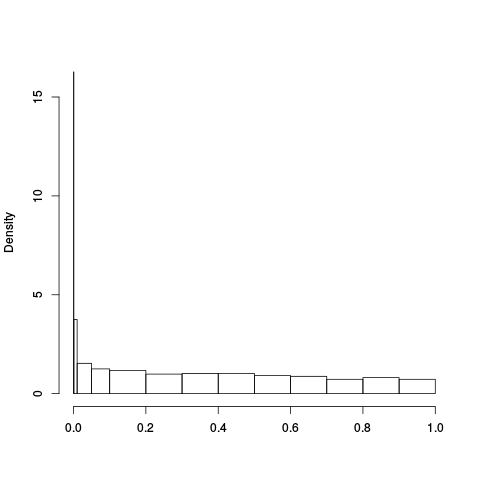 |
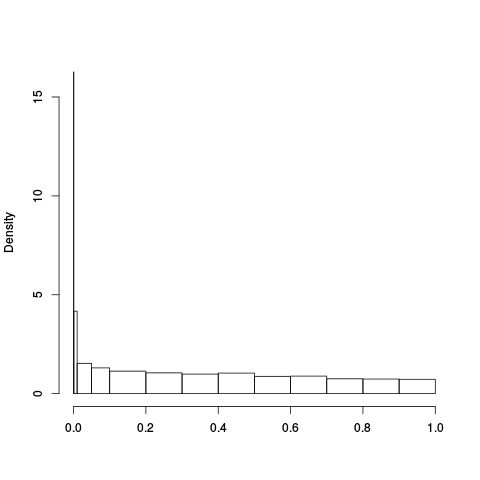 |
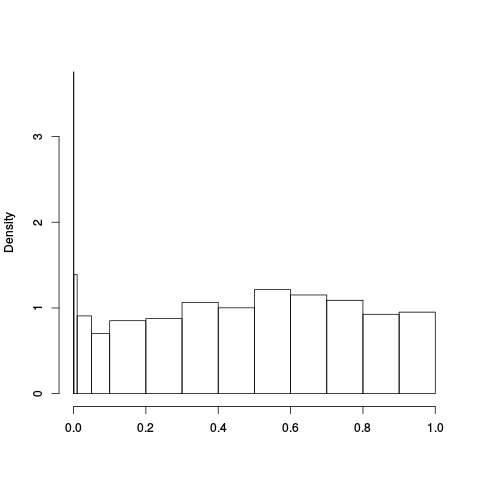 |
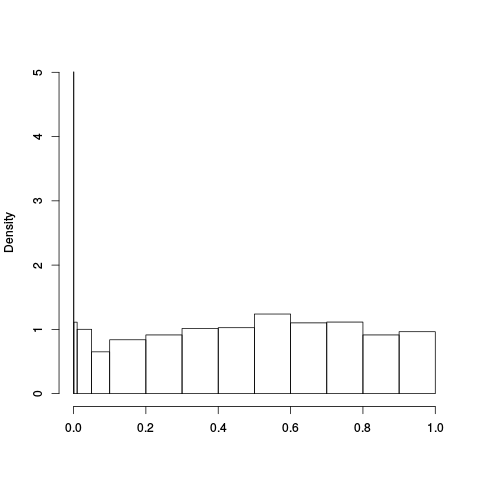 |
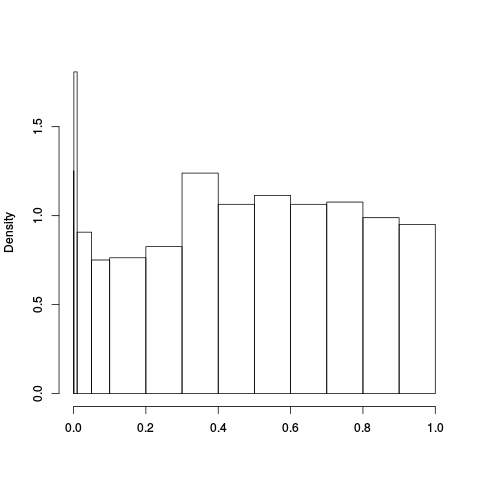 |
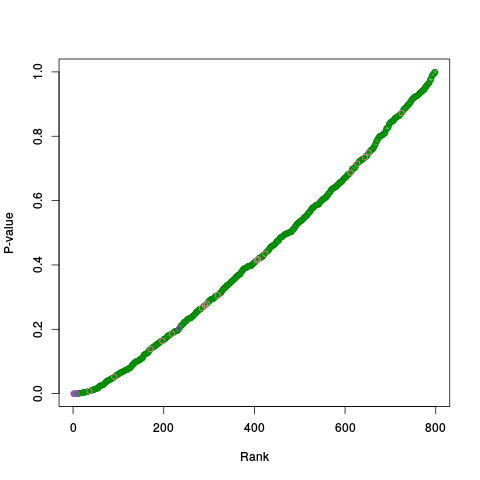 |
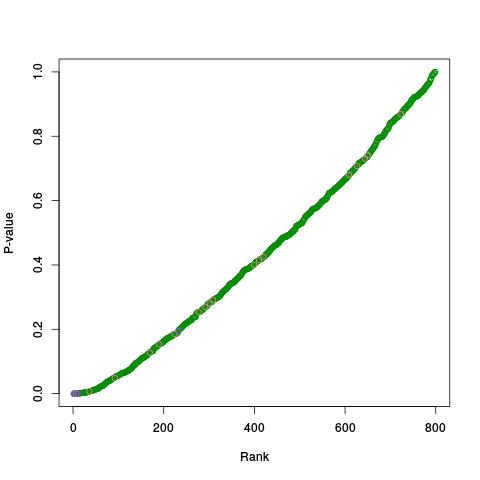 |
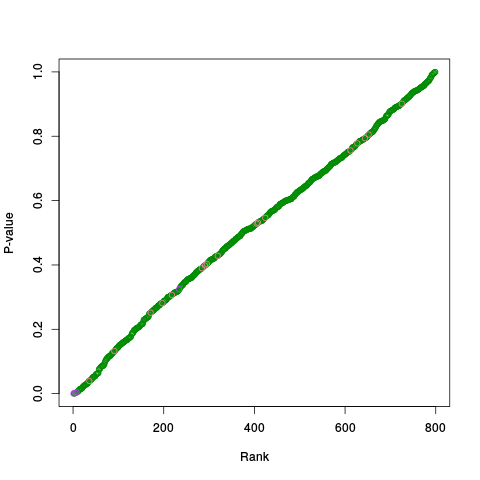 |
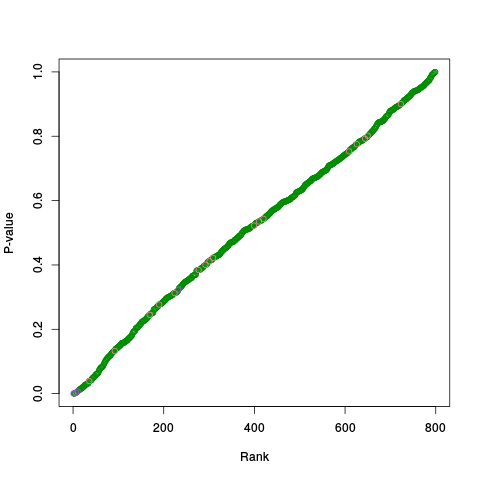 |
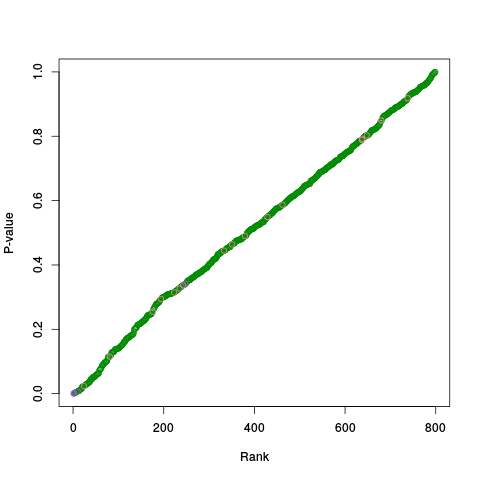 |
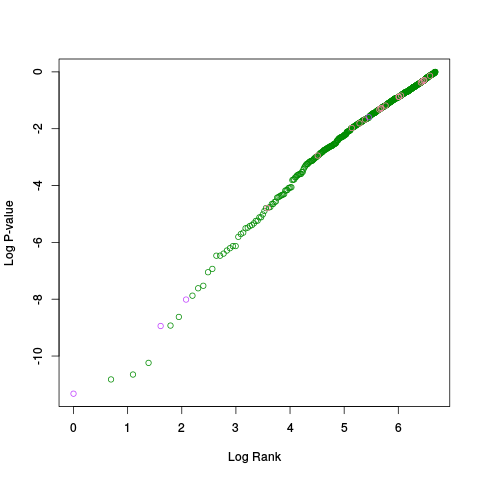 |
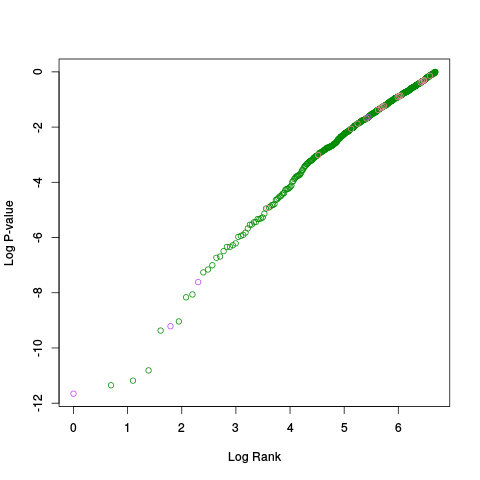 |
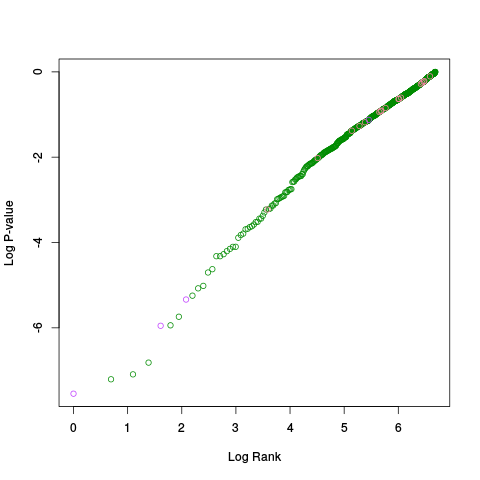 |
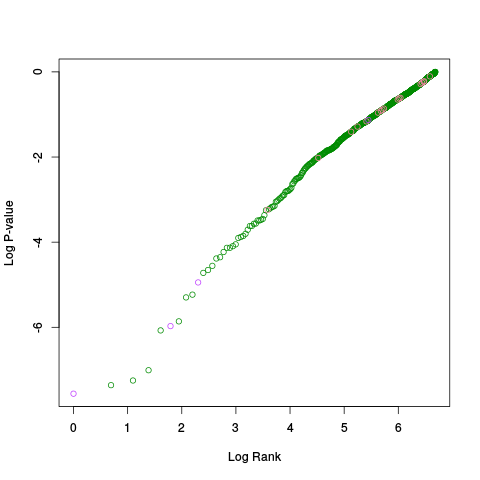 |
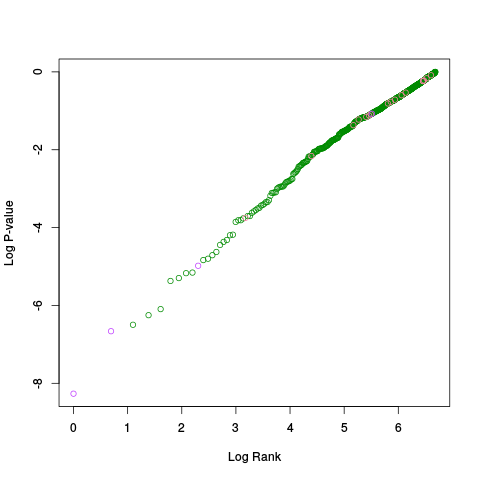 |
| Y | Y_c |
 |
 |
| standard | ebayes | rsvar | rsvar ebayes | evar |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
| standard | ebayes | rsvar | rsvar ebayes | evar |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
| Standard Coloring | standard | ebayes | rsvar | rsvar ebayes | evar |
 |
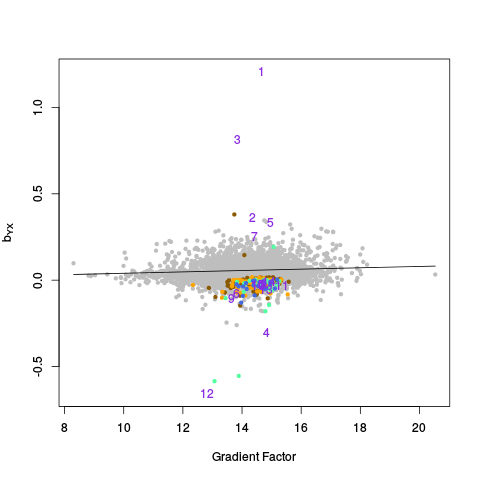 |
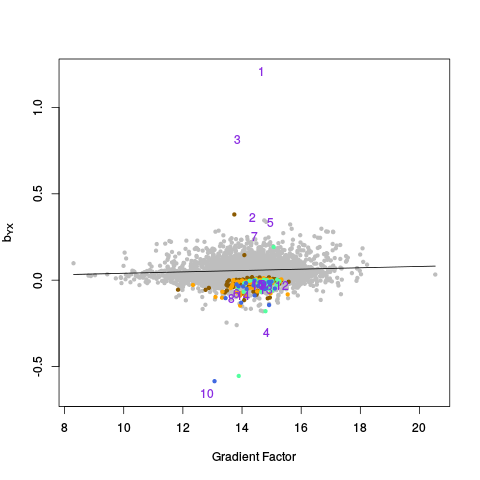 |
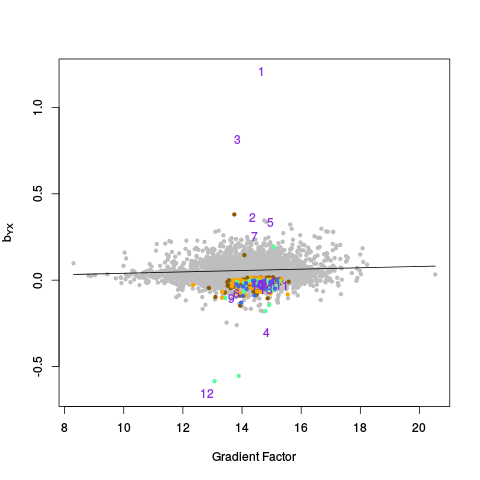 |
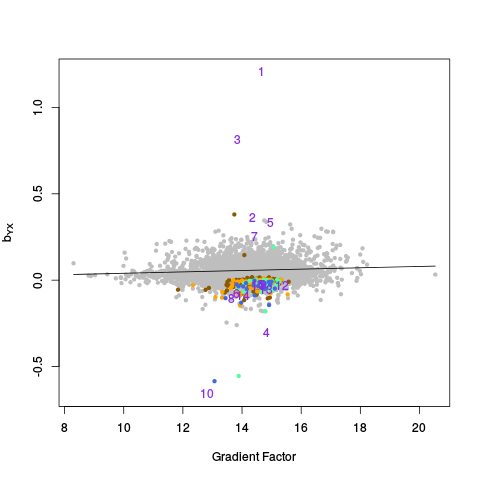 |
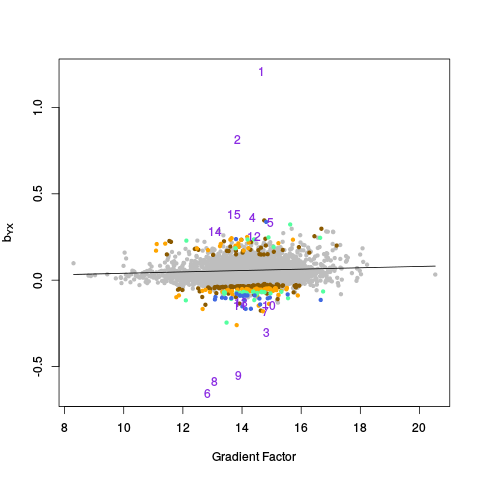 |
| Standard Coloring | standard | ebayes | rsvar | rsvar ebayes | evar |
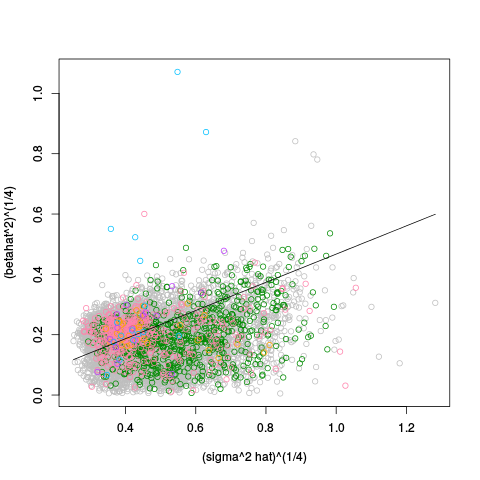 |
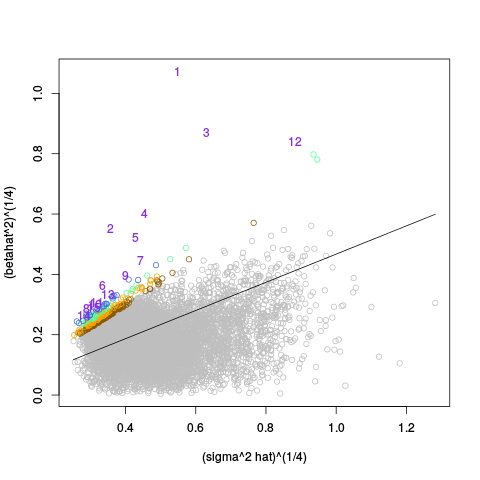 |
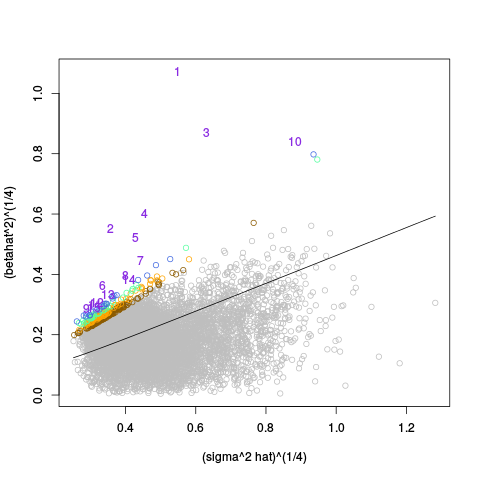 |
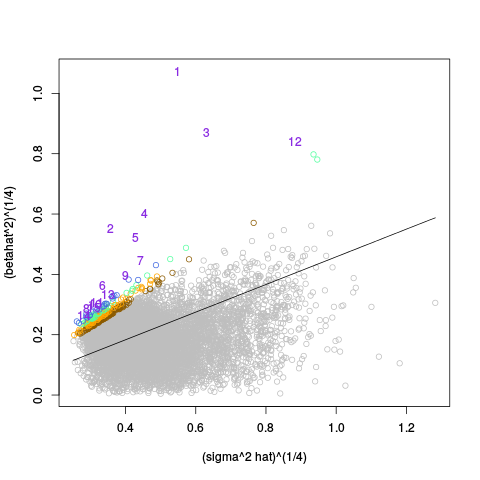 |
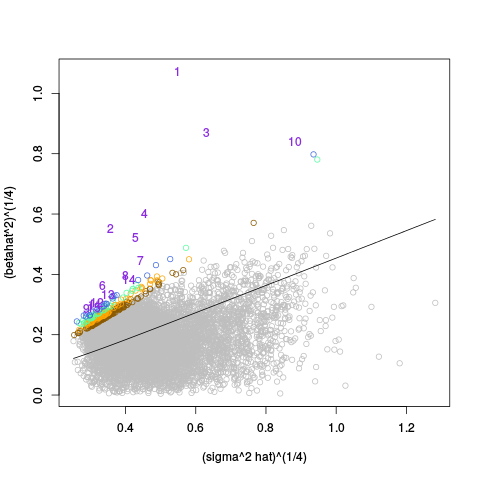 |
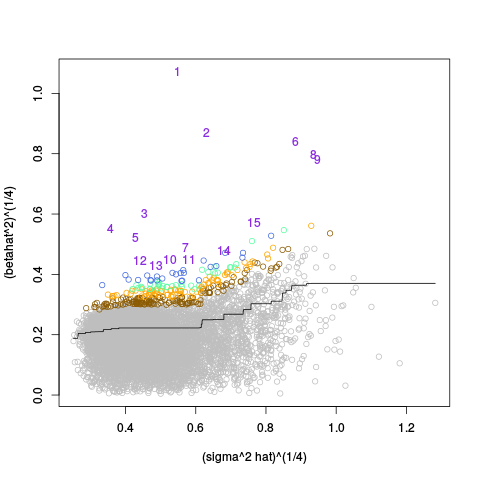 |
| standard | ebayes | rsvar | rsvar ebayes | evar | ||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
|
|
| Y | Y_c |
 |
 |
| standard | ebayes | rsvar | rsvar ebayes | evar |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
| standard | ebayes | rsvar | rsvar ebayes | evar |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
| Standard Coloring | standard | ebayes | rsvar | rsvar ebayes | evar |
 |
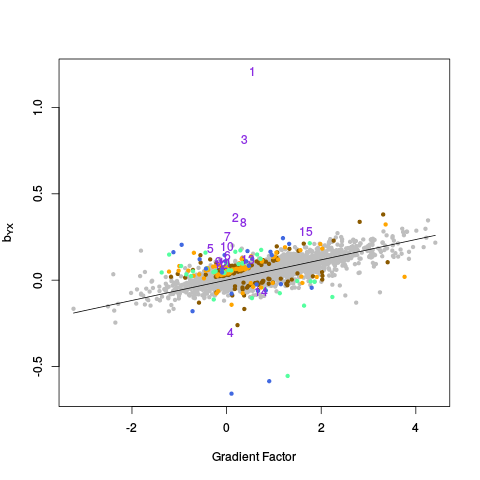 |
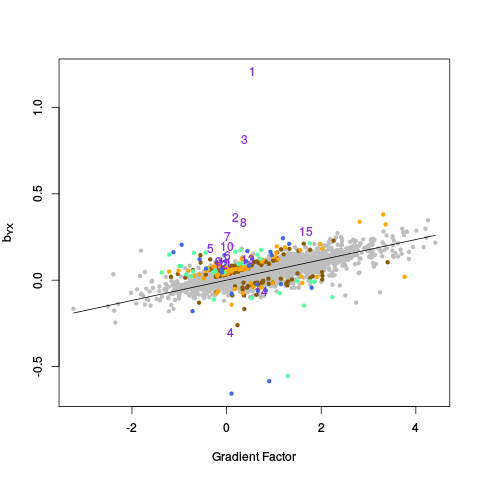 |
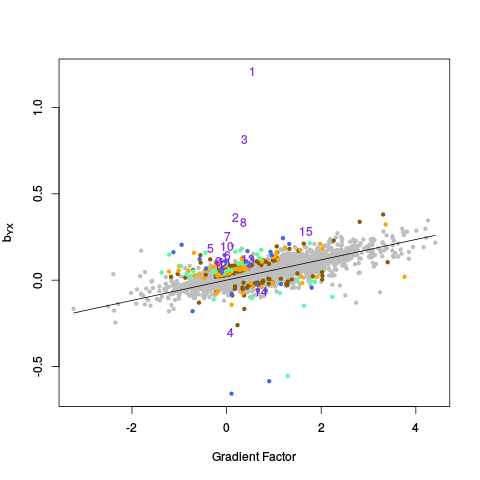 |
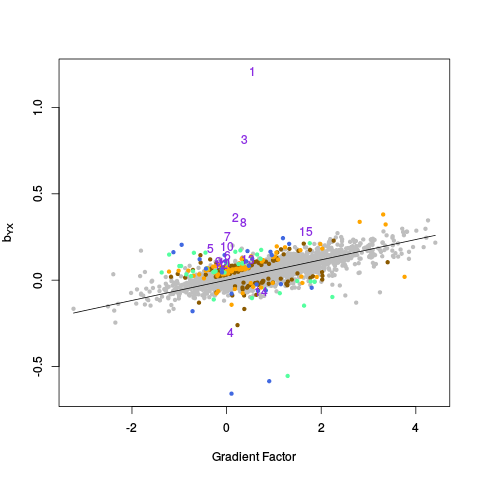 |
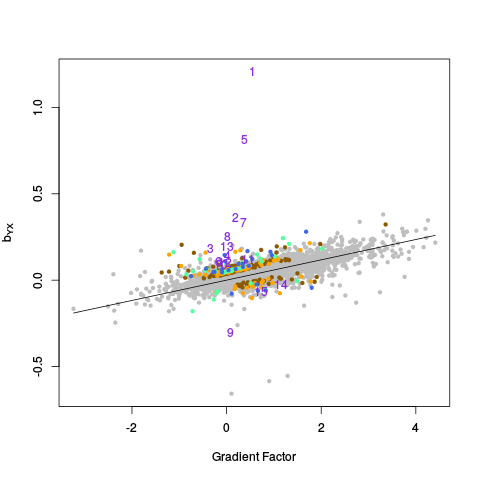 |
| Standard Coloring | standard | ebayes | rsvar | rsvar ebayes | evar |
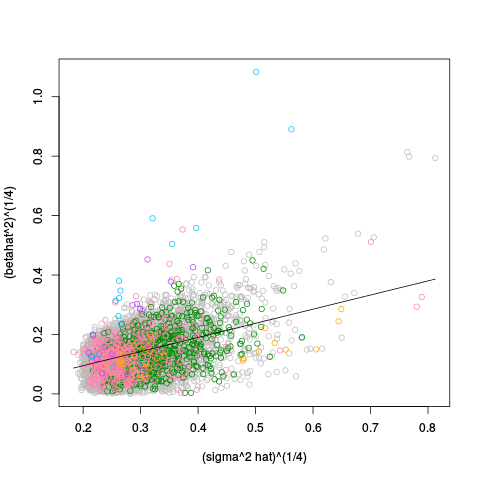 |
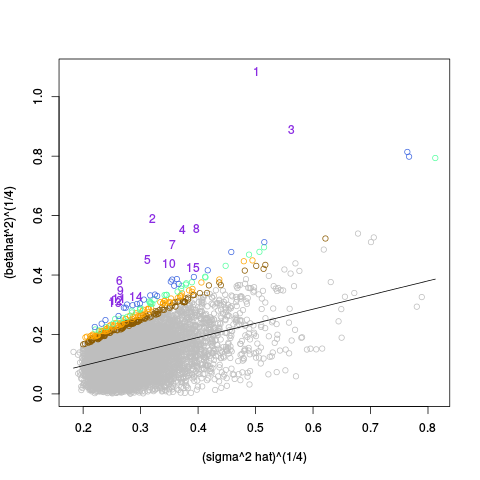 |
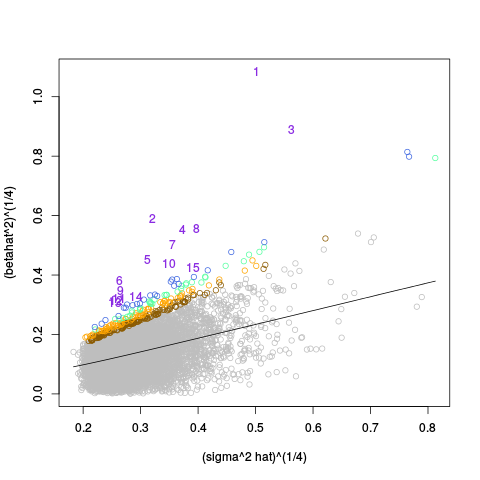 |
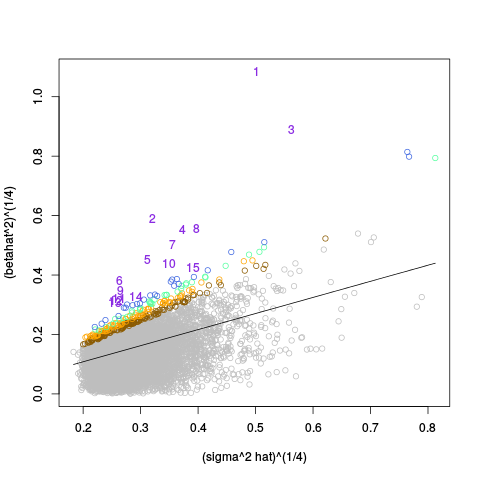 |
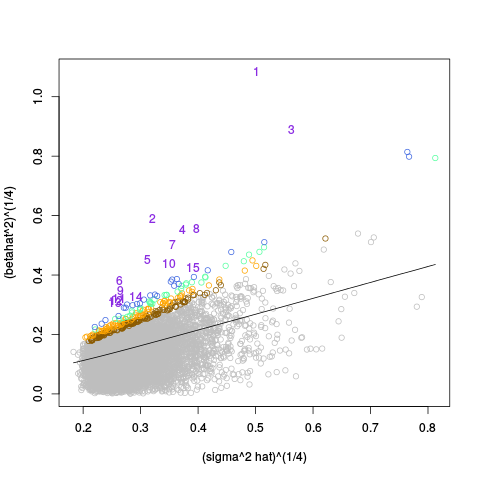 |
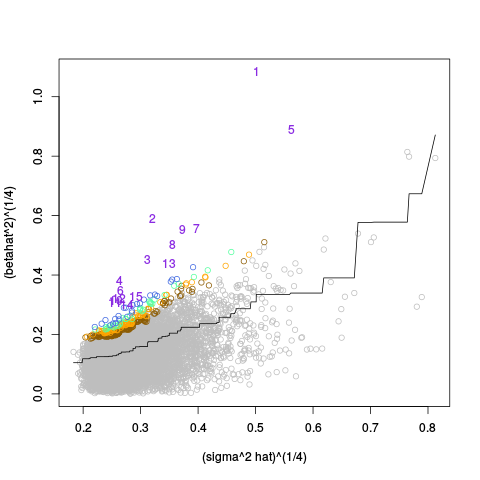 |
| standard | ebayes | rsvar | rsvar ebayes | evar | ||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
|
|
| standard | ebayes | rsvar | rsvar ebayes | evar | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
|
|
| 1 | 2 | 3 | 4 | 5 |
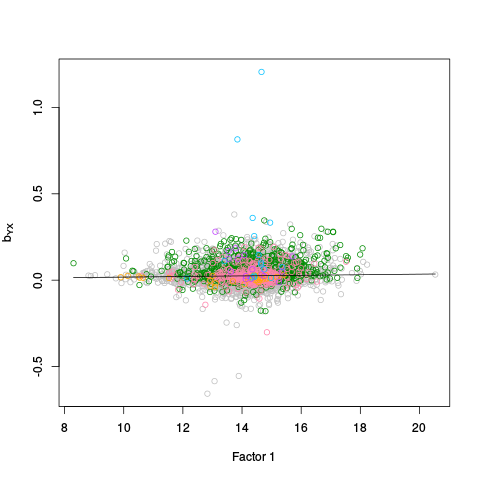 |
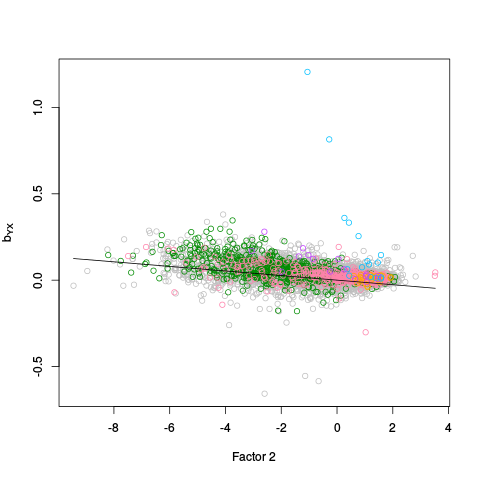 |
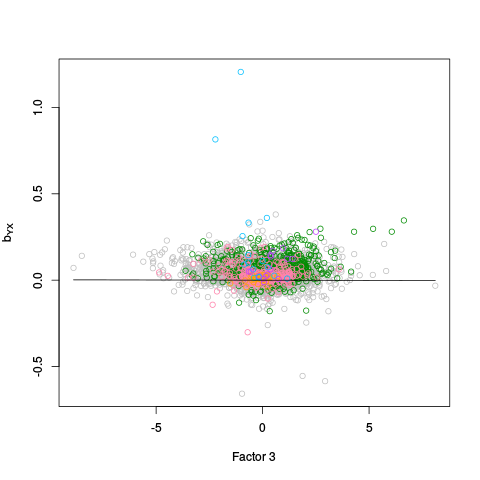 |
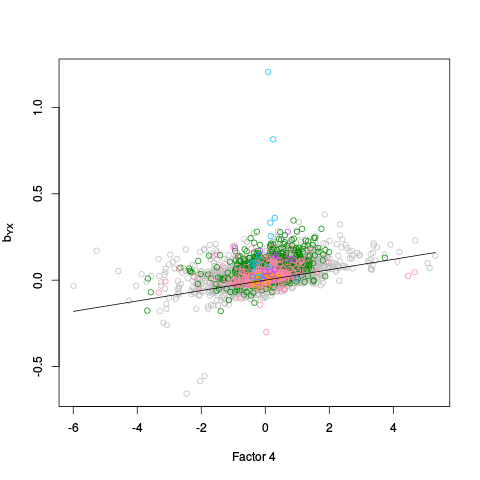 |
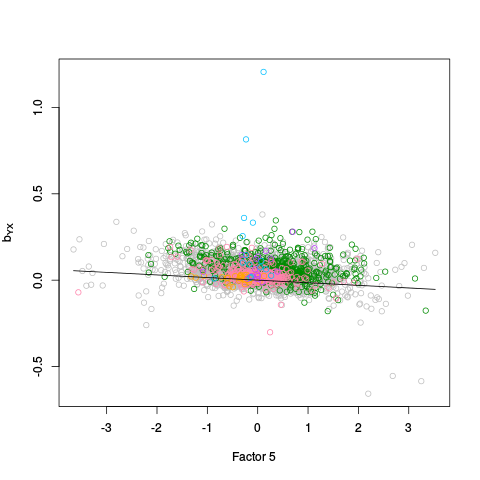 |
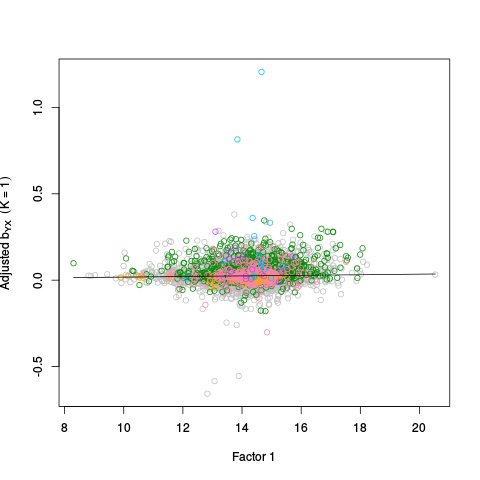 |
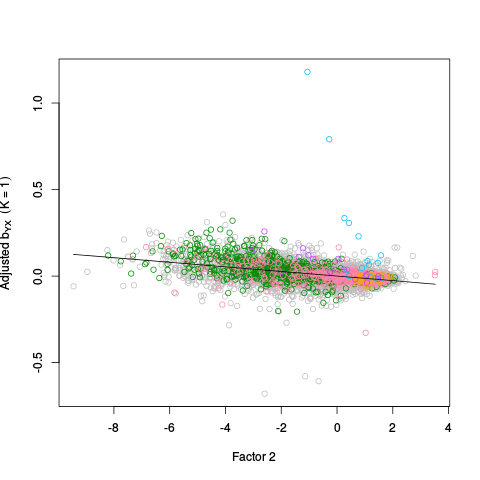 |
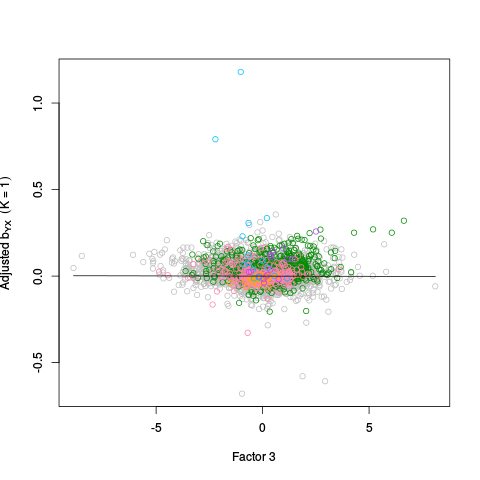 |
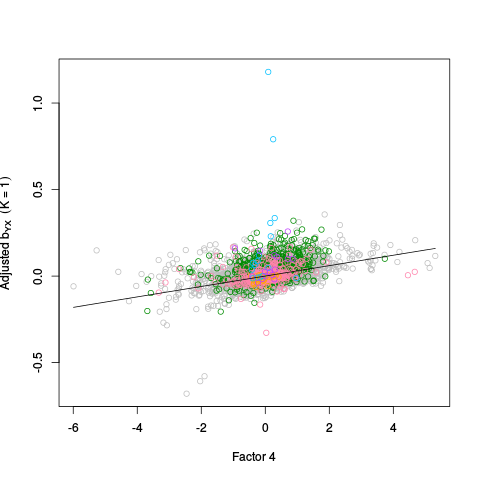 |
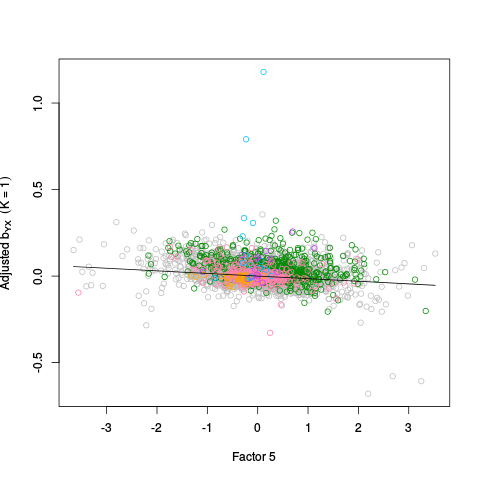 |
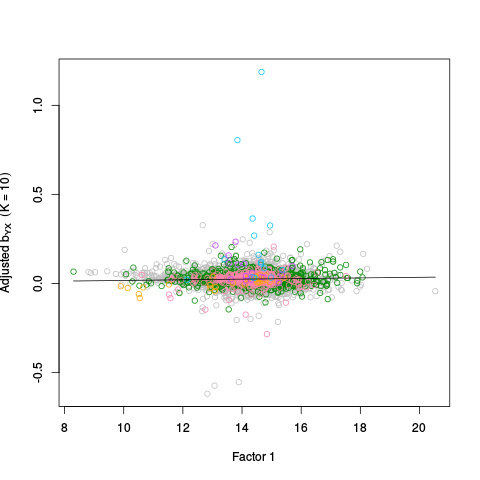 |
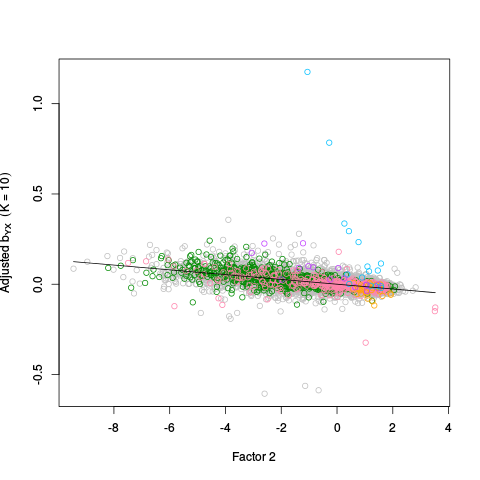 |
 |
 |
 |